蛋白互作网络PPI分析流程示例
蛋白互作网络(PPI, Protein-Protein Interaction Networks)可以查看基因之间的联系,进而锁定关键基因。一般使用STRING数据库获取蛋白互作信息。
STRING数据库:STRING: functional protein association networks (string-db.org)
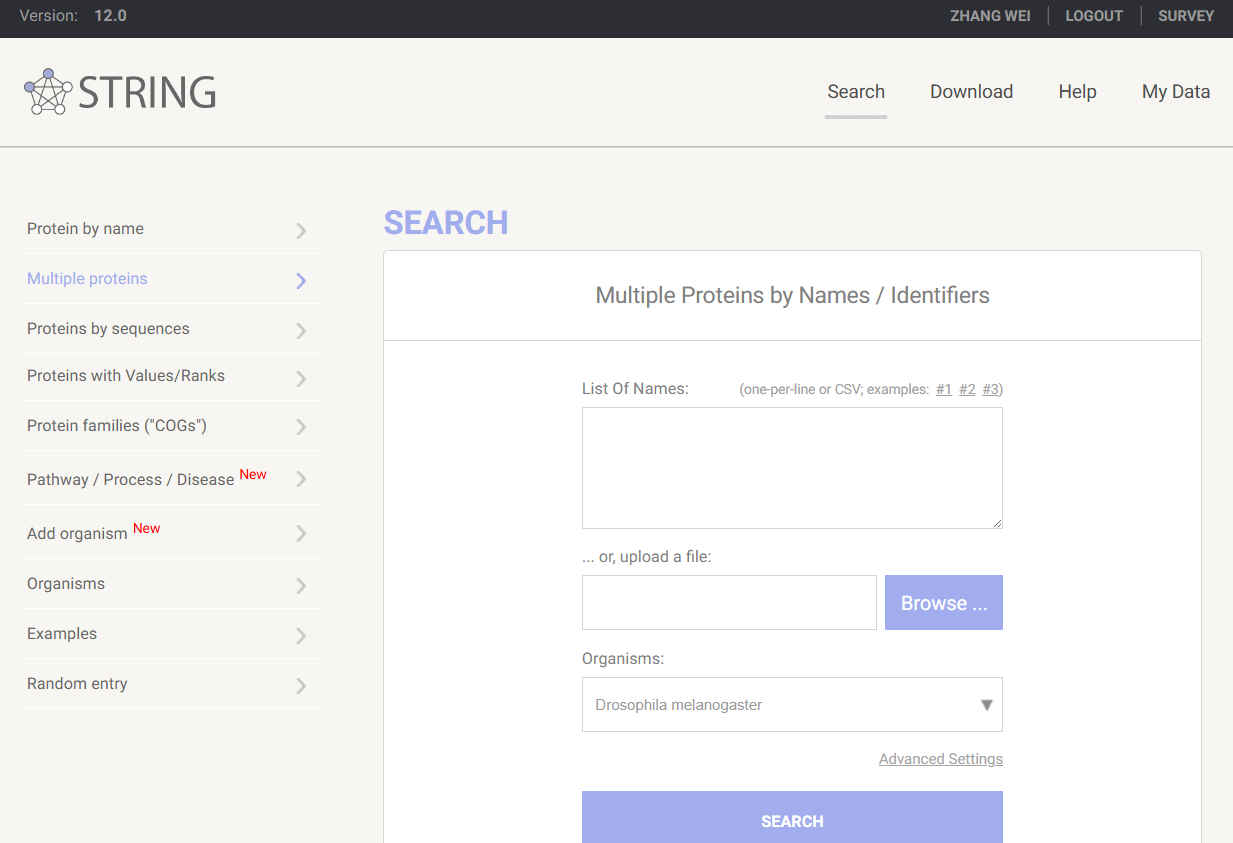
数据准备
要分析蛋白的名称列表,例如:
使用STRING数据库获取PPI结果
1. 选择Multiple proteins,将蛋白名称粘贴到List Of Names框中。我这里使用的是果蝇的相关蛋白,因此Organisms选择了Drosophila。点击search。
-
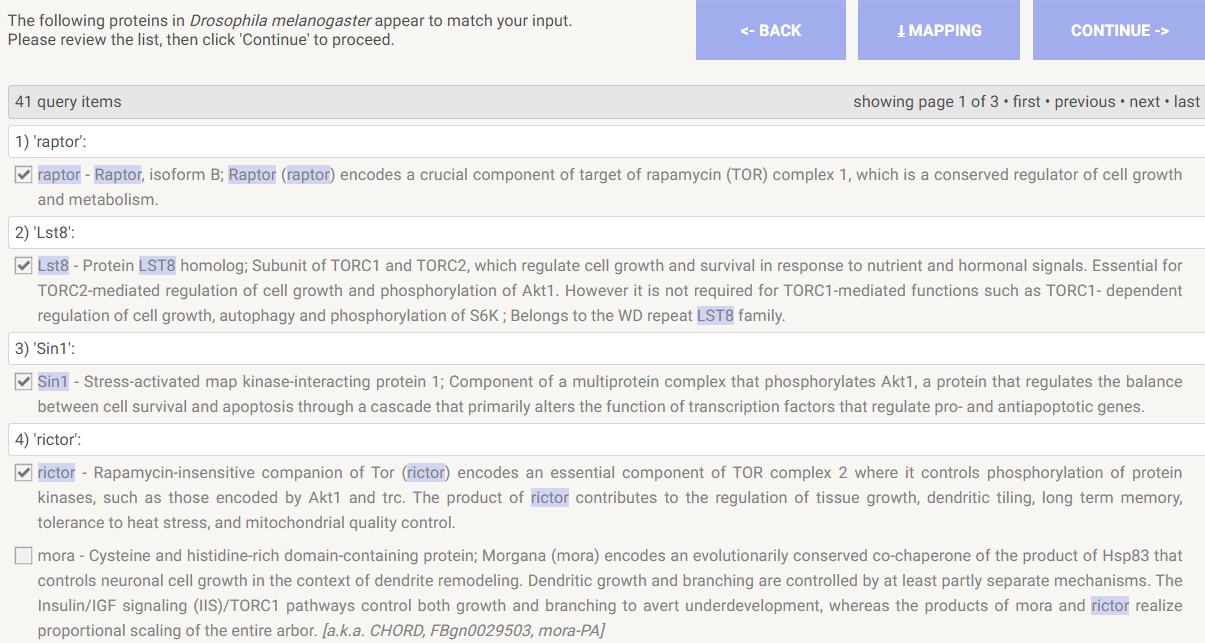
通过复选框选择对应的蛋白(默认选择的有时候不一定对),点击MAPPING可以下载当前查询到的蛋白信息,点击CONTINUE可以获得蛋白互作结果。
-
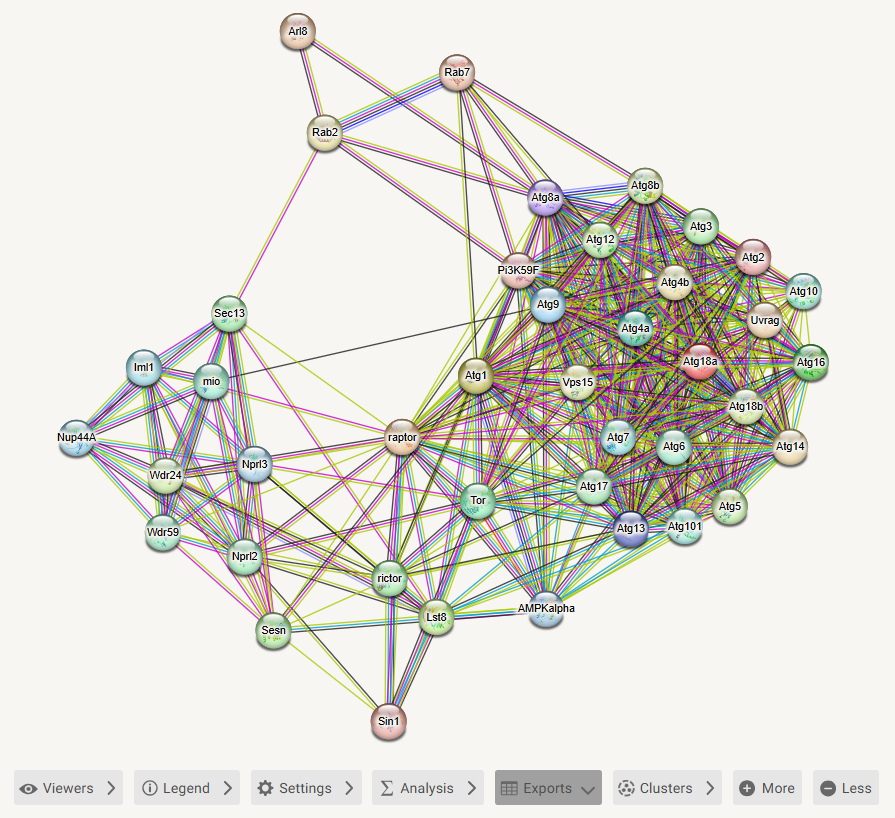
蛋白互作网络图如下
-
图例信息在Legend中

-
Settings中可以根据需要改变网络样式。
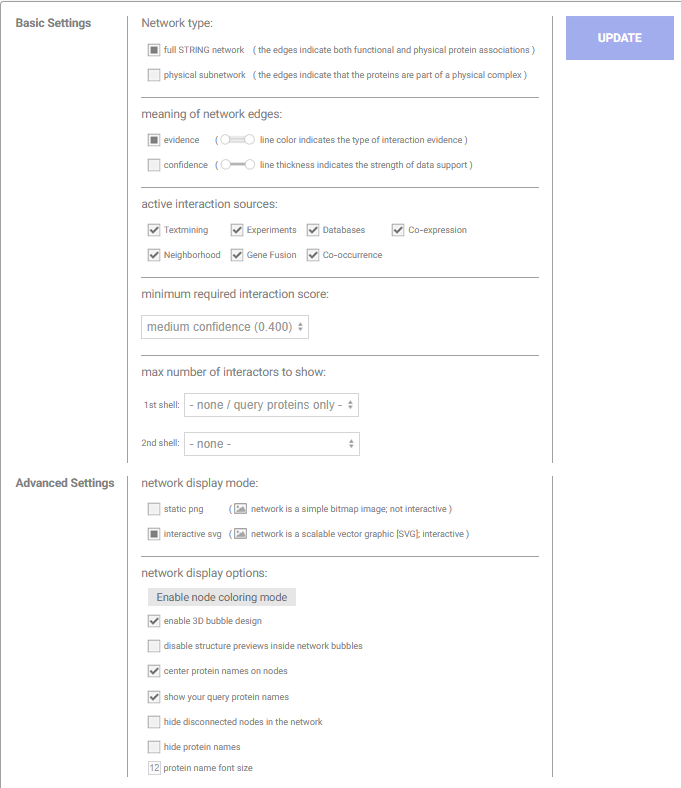
-
Exports可以将图片和蛋白互作信息导出。
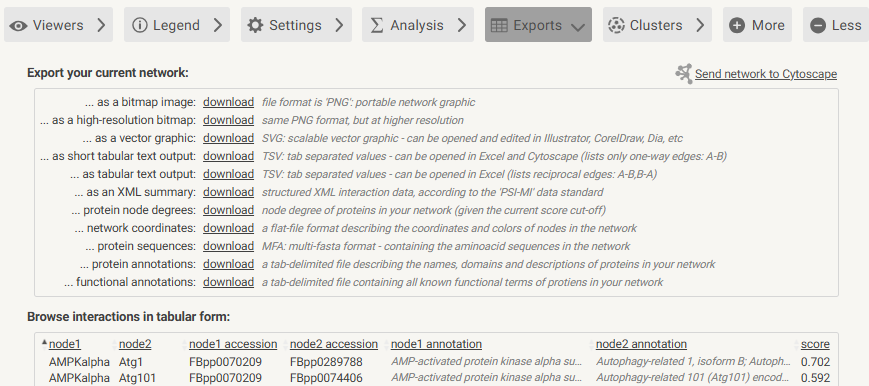
-
在网页界面,可以通过鼠标拖动改变蛋白分布。
-
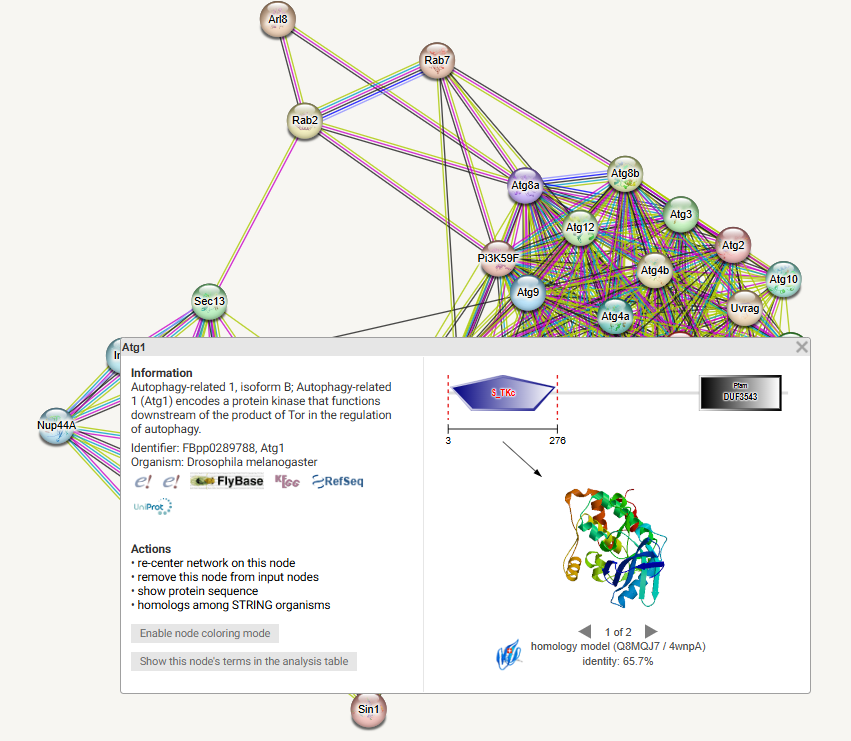
点击对应的蛋白节点能够查看蛋白结构信息。
![]()
-
点击连接线可以获得详细的连接数据
![]()
PPI(Protein-Protein Interaction,蛋白质-蛋白质相互作用)网络图是生物信息学中的一种图形表示,它展示了细胞内不同蛋白质之间的相互作用关系。
-
网络布局:
- 节点(Node):通常代表一个蛋白质。节点的大小可能表示蛋白质的度(与多少其他蛋白质相互作用)或者其他属性,如表达水平。
- 边(Edge):代表两个蛋白质之间的相互作用。边的粗细可能表示相互作用的强度或者置信度。
-
节点属性:
- 颜色:可以表示蛋白质的属性,如功能分类、亚细胞定位、是否为已知疾病相关蛋白等。
- 形状:不同的形状可以代表不同的蛋白质属性,如是否为酶、受体或其他类型。
-
网络特性:
- 连通性:高连通性的节点通常是网络中的关键蛋白,它们的缺失可能导致网络功能的显著变化。
- 模块性:网络中可能存在几个紧密连接的节点群,这些群被称为模块,可能代表特定的生物学过程。
-
功能分析:
- 使用GO(Gene Ontology)注释或其他功能数据库,对网络中的蛋白质进行功能富集分析,以识别网络中的主要生物学功能。
-
路径分析:
- 寻找网络中的关键路径,这些路径可能代表信号传导、代谢途径或其他生物学过程。
-
动态变化:
- 如果PPI网络图展示的是随时间或不同条件下变化的数据,分析这些动态变化可以帮助理解生物学过程如何响应外部刺激。
-
实验验证:
- PPI网络图通常是基于高通量实验数据(如酵母双杂交、质谱分析)构建的,因此实验验证是解读结果的关键步骤。