使用R语言从UniProt数据库检索蛋白信息-UniprotR包
UniprotR 包通过丰富的函数和可视化工具,使得从UniProt数据库中检索和分析蛋白质信息变得更加便捷和高效。
基本信息
-
版本:2.4.0
-
作者:Mohamed Soudy 和 Ali Mostafa
-
描述:连接到Uniprot,使用蛋白质的访问号检索信息,如名称、分类信息等。
-
URL:https://github.com/Proteomicslab57357/UniprotR
-
问题报告:https://github.com/Proteomicslab57357/UniprotR/issues
功能:
-
提供多种功能,包括构建基因树、位置树、转换ID、富集分析(包括生物学过程、细胞组分、KEGG、分子功能、REACTOME)、获取疾病关联、表达数据、家族域数据、一般信息、杂项信息、名称和分类信息、病理生物技术信息、PDB结构、蛋白质注释、功能、基因本体信息、蛋白质相互作用、蛋白质网络、蛋白质网络(全部)、蛋白质组信息、蛋白质组FASTA、PTM处理、出版物信息、序列长度、序列同源、序列、结构信息、亚细胞位置等。
-
提供了解析UniProt信息和绘制各种图表的功能,如GO分子功能、GO亚细胞定位、酸度、电荷、染色体信息、基因网络、GO全部、GO生物学过程、GO信息、GO术语、Gravy、理化性质、蛋白质存在状态、蛋白质GO生物、蛋白质GO细胞、蛋白质GO分子、蛋白质状态、蛋白质分类等。
图表和可视化:
-
除了数据检索,
UniprotR 还提供了多种数据可视化工具,帮助用户更直观地理解蛋白质的特性和功能。
安装和依赖:
-
该包不需要编译,可以直接从CRAN安装,且依赖于多个R包,方便用户进行数据分析和可视化。
安装
1 | install.packages("UniprotR") |
使用示例
GetAccessionList函数可用于从csv文件中获取UniProt Accession / s列表。
1 | library(UniprotR) |
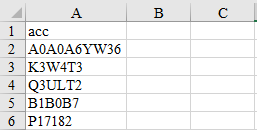
DataObjPath: Excel文件 (.csv) 的路径,文件内容如下图。
录入时间
1 | Dm_Acc_General <- GetGeneral_Information(Dm_Acc, "I:/Thesis/Analysis_Display/R_data") |
其他
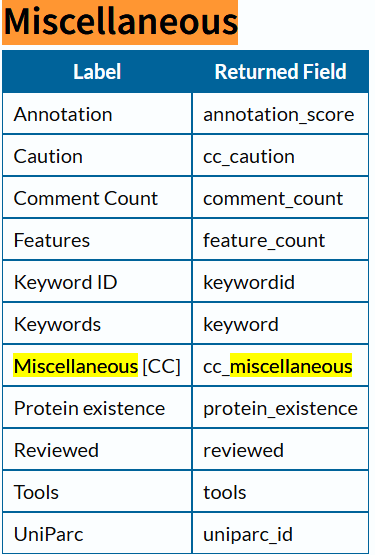
1 | Dm_Acc_Miscellaneous <- GetMiscellaneous(Dm_Acc, "I:/Thesis/Analysis_Display/R_data") |
获取分类信息
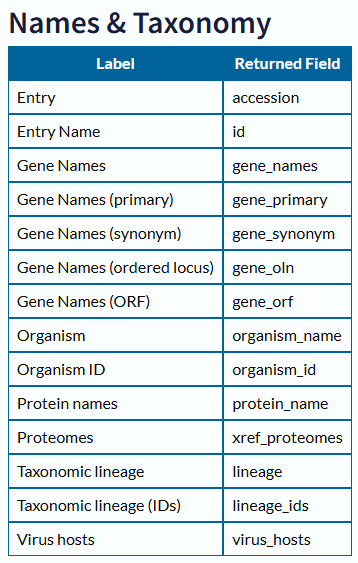
1 | TaxaObj <- GetNamesTaxa(Dm_Acc, "I:/Thesis/Analysis_Display/R_data") |
Pathology & Biotech
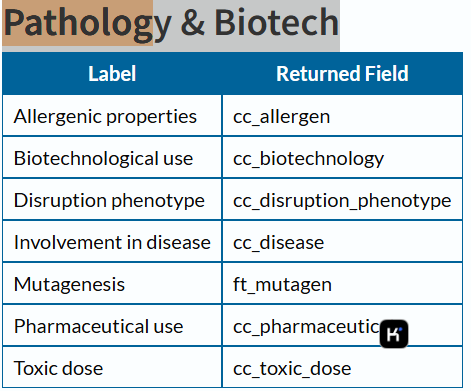
1 | Pathology_Biotech <- GetPathology_Biotech(Dm_Acc, "I:/Thesis/Analysis_Display/R_data") |
下载PDB结构数据
1 | Dm_pdbStructure <- GetpdbStructure(Dm_Acc, "I:/Thesis/Analysis_Display/R_data") |
蛋白功能
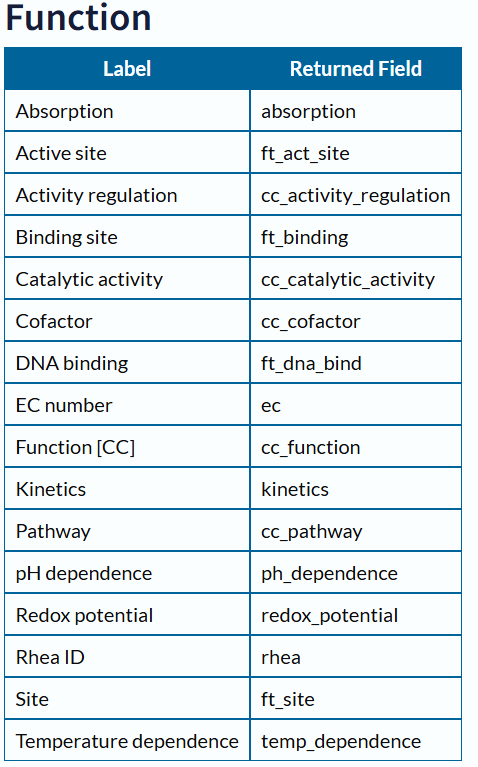
1 | Dm_ProteinFunction <- GetProteinFunction(Dm_Acc, "I:/Thesis/Analysis_Display/R_data") |
转换ID
将UniProtKB的AC / ID转换为UniProtKB中的任何数据库ID。详细信息参阅https://www.uniprot.org/help/id_mapping和https://raw.githubusercontent.com/MohmedSoudy/UniprotR/master/uniprot_ids.csv
1 | ConvertID(Dm_Acc,ID_from="UniProtKB_AC-ID",ID_to="OrthoDB",taxId="7227",path=NULL) |
ConvertID(ProteinAccList,ID_from="UniProtKB_AC-ID",ID_to=NULL,taxId=NULL,path=NULL)
ProteinAccList |
UniProt Accession/s 向量 |
ID_from |
数据库标识符的缩写,从中将Accession / ID进行转换。 |
ID_to |
数据库标识符缩写字符串,Accession / ID将转换为该字符串。默认为UniProtKB中可用的所有数据库标识符。见uniprot_ids.csv{.uri} |
taxId |
当ID_to为’ UniProtKB '时需要TaxId字符串。例如 ‘9606’ for human。 |
path |
保存excel文件的路径。 |
获取蛋白序列-fasta格式
通过 UniProt 中的蛋白ID,下载蛋白序列。
1 | # 下载UniProt的蛋白序列并保存为fasta文件 |
蛋白序列信息
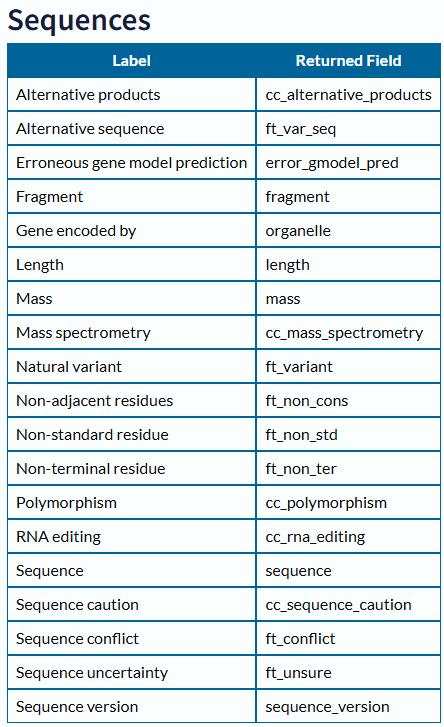
1 | # 获取序列信息 |
蛋白互作-STRING
连接到stringdb并检索输入列表之间的PPI
GetproteinNetwork_all(ProteinAccList , directorypath = NULL, SpeciesID = 7227)
1 | # 根据导入的数据生成PPI |
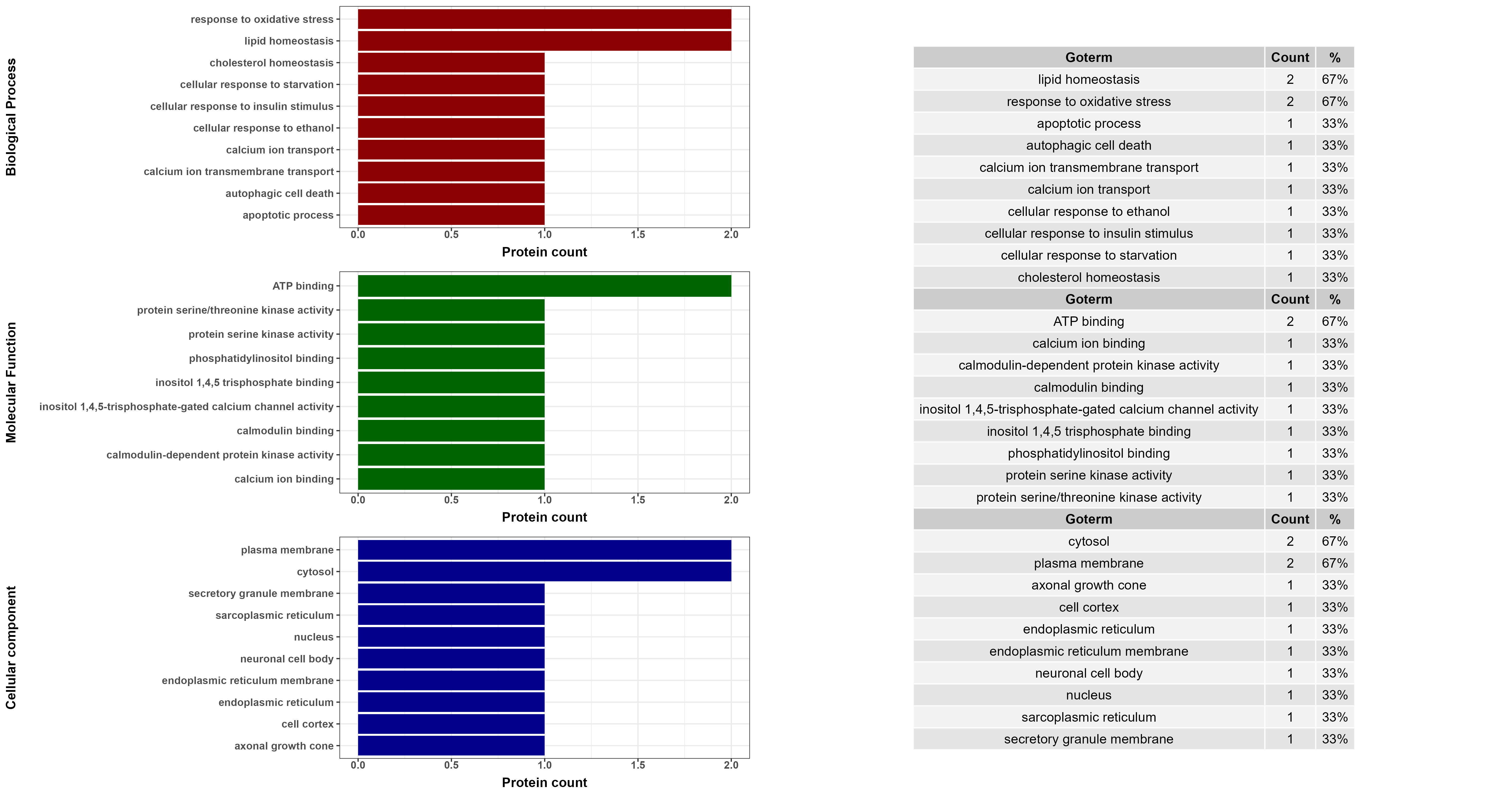
GO分析-“Gene ontology”(基因本体论)
格式
PlotGOBiological(GOObj, Top = 10, directorypath = NULL)
Plot.GOMolecular(GOObj, Top = 10, directorypath = NULL)
Plot.GOSubCellular(GOObj, Top = 10, directorypath = NULL)
PlotGoInfo(GOObj , directorypath = NULL)
PlotGOAll(GOObj, Top = 10, directorypath = NULL, width = width, height = height)
GOObj 参数: 指的是得自 GetProteinGOInfo 函数的数据集。
1 | # 获取Gene ontolgy信息 |
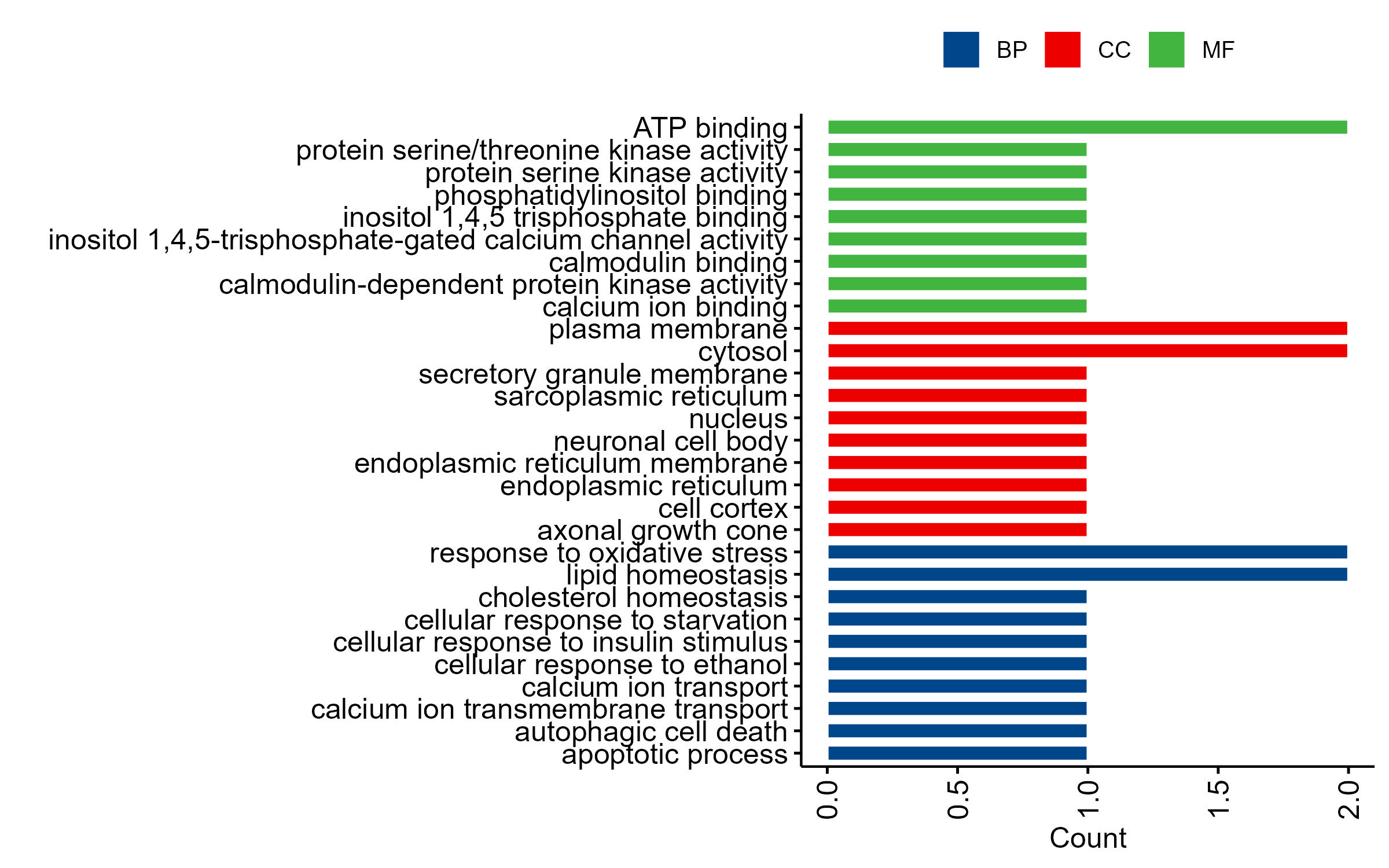
KEGG富集分析
1 | Enrichment.KEGG(Dm_Acc,OS="dmelanogaster",p_value=0.05,directorypath="I:/Thesis/Analysis_Display/R_data",top=10) |
蛋白表达信息
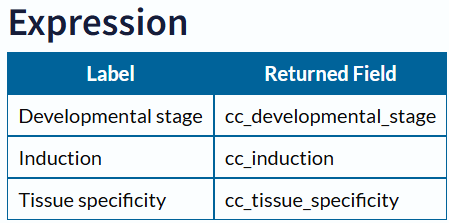
1 | Dm_Acc_Expression <- GetExpression(Dm_Acc, "I:/Thesis/Analysis_Display/R_data") |
蛋白结构信息
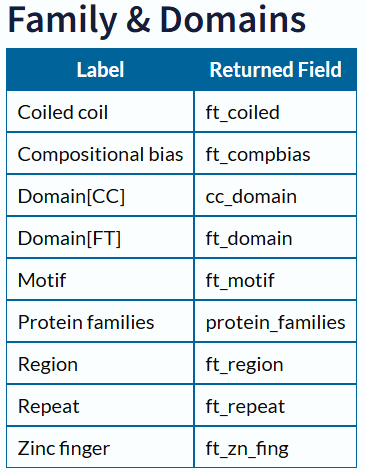
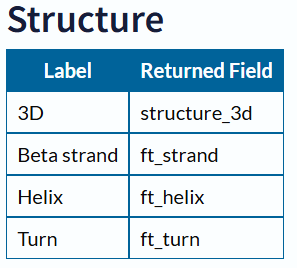
1 | # 蛋白二级结构 |
亚细胞定位
1 | GetSubcellular_location(Dm_Acc, "I:/Thesis/Analysis_Display/R_data") |
发表的文献
1 | GetPublication(Dm_Acc, "I:/Thesis/Analysis_Display/R_data") |

其他R包-UniProt
drawProteins包
根据从Uniprot蛋白质数据库中获得的数据绘制蛋白结构示意图。
其基本工作流程是:
-
提供一个或多个Uniprot ID;
-
从Uniprot API中获取特征列表;
-
绘制这些蛋白质的基本链;
-
根据需要添加特征。
drawProteins使用包httr与Uniprot API进行交互,并将一个JSON对象提取到R中。JSON对象用于创建数据表。 然后使用ggplot2绘制蛋白示意图。 详细见 https://www.bioconductor.org/packages/release/bioc/vignettes/drawProteins/inst/doc/drawProteins_BiocStyle.html
queryup包
从’ UniProtKB ’ REST API中检索蛋白质信息
参考
-
Proteomicslab57357/UniprotR: Retrieving Information of Proteins from Uniprot
-
CRAN: Package UniprotR
-
UniprotR: Retrieving and visualizing protein sequence and functional information from Universal Protein Resource (UniProt knowledgebase) - PubMed
-
自学R语言–使用UniprotR包提取Uniprot数据库内容 - 知乎

